大球蓋菇是近年來全國范圍內產量急劇攀升的食用菌種類,市場前景廣闊。針對傳統菌株鑒定手段準確率低、重復性差、分辨力有限等問題,并充分滿足產業對品種、專利菌株精準鑒定和知識產權保護技術的迫切需求,近日,中國科學院微生物研究所趙瑞琳團隊聯合中國農業科學院農業資源與農業區劃研究所高巍副研究員等研究人員,在Microorganisms期刊發表題為《High-Resolution Core Gene-Associated Multiple Nucleotide Polymorphism (cgMNP) Markers for Strain Identification in the Wine Cap Mushroom Stropharia rugosoannulata》的研究論文。該研究成功將基因組范圍內的核心基因相關多核苷酸多態性(cgMNP)標記應用于大球蓋菇,實現了菌株的高分辨率分型,標志著cgMNP作為“第二代MNP分子標記”在真菌領域的進一步拓展。
本研究是在大球蓋菇中首次成功驗證cgMNP方法,對105個大球蓋菇菌株基因組重測序,鑒定276萬SNP,篩選83個核心基因的cgMNP標記。基于cgMNP構建的系統發育樹與遺傳相似性分析,不僅實現了菌株的高分辨率區分,還揭示了譜系劃分與馴化分化的遺傳基礎。特別地,研究發現線粒體脂代謝相關核心基因 Phosphatidate cytidylyltransferase mitochondrial 內含865個多態性位點,單基因即可實現全部85個栽培菌株的區分,表現出極高的物種內部分辨能力,具有單基因快速溯源與品種認證的潛力。
該成果是團隊自2022年香菇和金針菇多核苷酸多態性(MNP)為基礎的第一代菌株鑒定標記(Ling et al., Mycosphere,2022;Liu et al., Journal of Fungi, 2023)開發;2024年,團隊在雙孢蘑菇中首創cgMNP方法(Liu et al., Microbial Biotechnology)后,實現了以核心基因為錨點的通用型分子標記開發的又一個成功案例。實現了從特定物種的專屬性MNP標記開發,到基于核心基因的cgMNP標記實現多物種通用鑒定,再到依托高多態性核心基因開展單基因快速鑒定的技術迭代,開啟了真菌品種分型與知識產權保護的新路徑。
中國科學院微生物所劉飛助理研究員為論文第一作者,中國農業科學院農業資源與農業區劃研究所副研究員高巍和微生物所趙瑞琳研究員為論文共同通信作者,河北農業大學李守勉教授團隊也參與了部分工作。
本研究得到國家重點研發計劃(2022YFD1200605)、北京食用菌創新團隊(BAIC03)和河北省重點研發(21326315D)等項目的資助。
全文鏈接:https://www.mdpi.com/2076-2607/13/7/1685
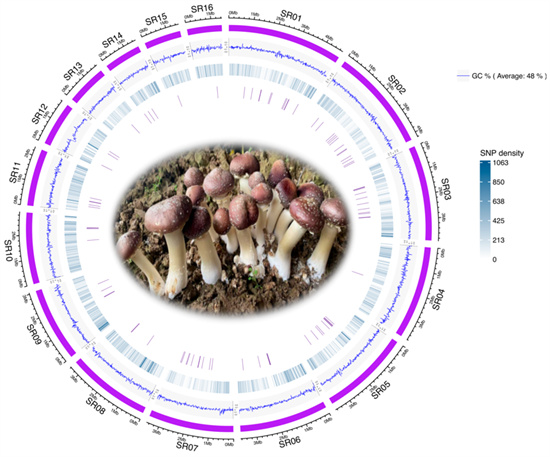
圖1.大球蓋菇基因組圈圖
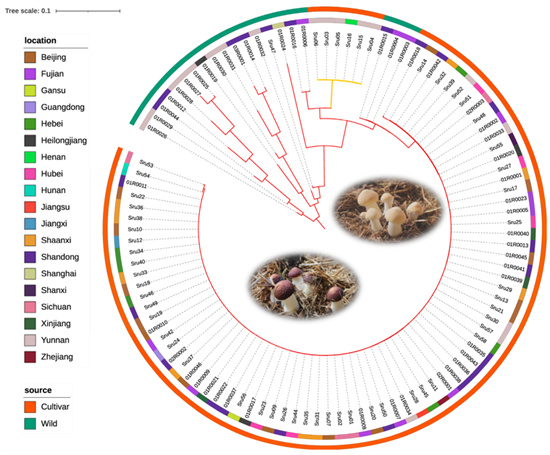
圖2.基于核心基因相關MNP構建的大球蓋菇系統發育樹
參考文獻:
1. Liu, F.; Cao, B.; Dai, H.; Li, G.; Li, S.; Gao, W.; Zhao, R. High-Resolution Core Gene-Associated Multiple Nucleotide Polymorphism (cgMNP) Markers for Strain Identification in the Wine Cap Mushroom Stropharia Rugosoannulata. Microorganisms 2025, 13, 1685, doi:10.3390/microorganisms13071685.
2. Liu, F.; Cai, Z.-X.; Kang, W.-Y.; Chen, W.-Z.; Lu, Y.-P.; Chen, M.-Y.; Zhao, R.-L. A New Method for Constructing High-Resolution Phylogenomic Topologies Using Core Gene-Associated MNP Markers: A Case Study From Agaricus Bisporus. Microbial Biotechnology 2025, 18, e70070, doi:10.1111/1751-7915.70070.
3. Liu, F.; Wang, S.-H.; Jia, D.-H.; Tan, H.; Wang, B.; Zhao, R.-L. Development of Multiple Nucleotide Polymorphism Molecular Markers for Enoki Mushroom (Flammulina Filiformis) Cultivars Identification. JoF 2023, 9, 330, doi:10.3390/jof9030330.
4. Ling, Y.; Zhang, M.; Ling, Z.; Zhao, Z. Evolutionary Relationship and a Novel Method of Efficient Identification of Lentinula Edodes Cultivars in China. Mycosphere 2022, 13, 56–85.