9月28日,華中農(nóng)業(yè)大學(xué)植物科學(xué)技術(shù)學(xué)院邊銀丙教授領(lǐng)銜的應(yīng)用真菌研究團(tuán)隊(duì)在Journal of Advanced Research(2020 IF=10.479)上在線發(fā)表了題為“Population genomics provides insights into the genetic basis of adaptive evolution in the mushroom-forming fungus Lentinula edodes”的研究論文,張荊城碩士和博士生沈楠為論文共同第一作者,肖揚(yáng)副教授為通訊作者。該研究首次使用群體基因組方法探究了具有重要經(jīng)濟(jì)價(jià)值的大型真菌進(jìn)化史,解析了香菇適應(yīng)性進(jìn)化的遺傳基礎(chǔ),將為香菇優(yōu)良品種選育奠定重要的理論基礎(chǔ)。
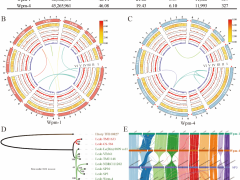
香菇是全球最重要的食用菌,產(chǎn)量占世界栽培食用菌總產(chǎn)量的22%,居各種類首位。生態(tài)適應(yīng)和人工選擇都可推動(dòng)香菇基因組的進(jìn)化,但其表型-基因型-適應(yīng)性之間的聯(lián)系尚不明確。研究團(tuán)隊(duì)基于133個(gè)香菇菌株,開展了基因組重測(cè)序和栽培試驗(yàn)。群體基因組研究將供試菌株分為3個(gè)亞群,亞群間具有顯著的地理分布特征、表型分化和溫度響應(yīng)差異。3個(gè)亞群獨(dú)立分化于36,871世代以前,現(xiàn)代香菇栽培品種可能起源于我國東北附近地區(qū)。結(jié)合基因組掃描和全基因組關(guān)聯(lián)分析,研究團(tuán)隊(duì)發(fā)現(xiàn)了大量與群體間遺傳和表型分化相關(guān)的候選基因。其中環(huán)境響應(yīng),信號(hào)轉(zhuǎn)導(dǎo),轉(zhuǎn)錄調(diào)控,細(xì)胞周期調(diào)控,真菌細(xì)胞壁重塑,蛋白質(zhì)降解,以及代謝和運(yùn)輸相關(guān)功能基因尤為重要。結(jié)合子實(shí)體發(fā)育的轉(zhuǎn)錄組研究發(fā)現(xiàn),相當(dāng)一部分群體分化相關(guān)基因參與了子實(shí)體發(fā)育的過程。研究團(tuán)隊(duì)構(gòu)建了香菇適應(yīng)性進(jìn)化過程中遺傳和表型分化的模型。研究表明,對(duì)當(dāng)?shù)丨h(huán)境尤其是溫度的適應(yīng)引發(fā)了香菇群體的遺傳和表型分化。
研究團(tuán)隊(duì)還結(jié)合基因組、遺傳和表型數(shù)據(jù)解析了香菇子實(shí)體相關(guān)性狀的遺傳基礎(chǔ),深化了對(duì)蘑菇形成真菌子實(shí)體發(fā)育的認(rèn)識(shí)水平。今年3月論文以“RNA-Seq-based high-resolution linkage map reveals the genetic architecture of fruiting body development in shiitake mushroom, Lentinula edodes”為題,在線發(fā)表于Computational and Structural Biotechnology Journal雜志(2020 IF=7.271)。研究團(tuán)隊(duì)構(gòu)建了香菇的超高密度遺傳連鎖圖,定位了29個(gè)與子實(shí)體相關(guān)性狀關(guān)聯(lián)的QTLs, 以及3個(gè)基因組熱點(diǎn)區(qū)域。利用meta-QTL分析檢測(cè)到7個(gè)多效性QTLs,發(fā)現(xiàn)它們調(diào)控子實(shí)體相關(guān)性狀間的表型,挖掘出33個(gè)調(diào)控子實(shí)體發(fā)育的重要候選基因。張琳碩士和龔文兵博士為Computational and Structural Biotechnology Journal文章的第一作者,通訊作者為肖揚(yáng)副教授。
香港中文大學(xué)關(guān)海山教授團(tuán)隊(duì),美國克拉克大學(xué)David Hibbett教授團(tuán)隊(duì)及美國能源部聯(lián)合基因組研究所Igor Grigoriev教授團(tuán)隊(duì)參與了部分研究工作。相關(guān)研究獲得了國家自然科學(xué)基金、國家重點(diǎn)研發(fā)計(jì)劃和湖北省重點(diǎn)研發(fā)計(jì)劃等項(xiàng)目的資助。
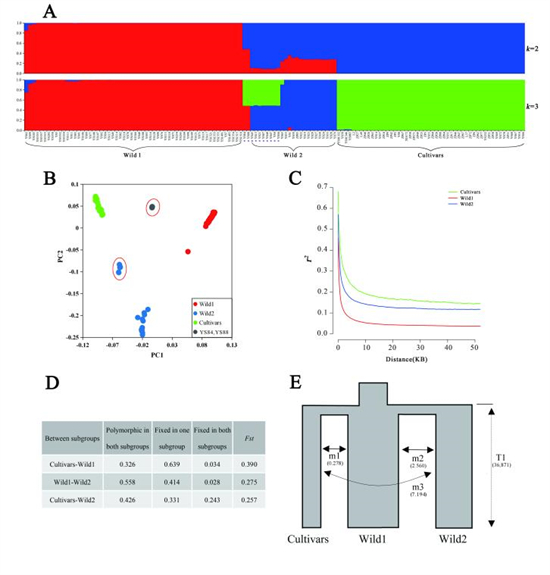
圖1 香菇群體結(jié)構(gòu)與分化
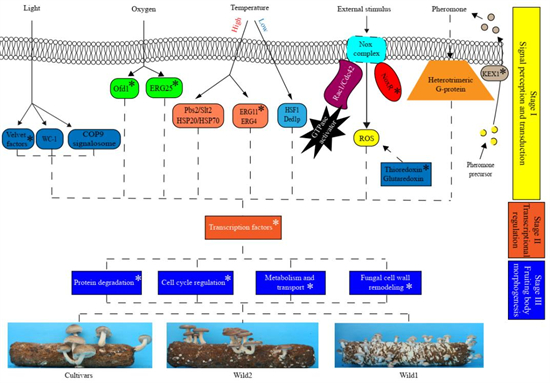
圖2 適應(yīng)進(jìn)化過程中香菇遺傳和表型分化模型
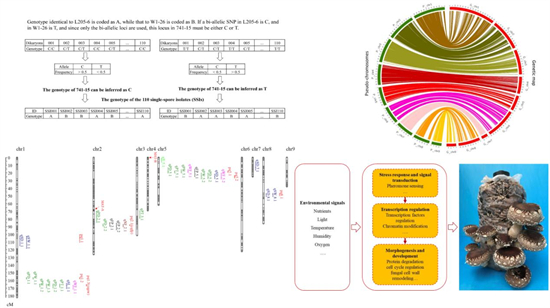
圖3 香菇子實(shí)體相關(guān)性狀的遺傳基礎(chǔ)解析
文章鏈接:https://www.sciencedirect.com/science/article/pii/S2090123221001892
文章鏈接:https://www.sciencedirect.com/science/article/pii/S200103702100088X?via%3Dihub